Decision Trees no R: Análise do ENEM 2018
Olá! No post de hoje, utilizarei uma ferramenta fascinante para predição e explicação em modelos tanto de regressão quanto de classificação: as árvores de decisão, ou decision trees.
As árvores de decisão, como no exemplo abaixo, são traçadas a partir de uma combinação de nós e folhas, sendo cada nó uma variável com um determinado valor ou categoria dividida e cada folha com um valor ou categoria predita. Tal recurso é interessante pois a interpretação é facilitada pelo apelo gráfico, e nos permite entender quais variáveis são importantes e determinantes na predição/explicação de um modelo.

Exemplo de Decision Tree
Há algum tempo, vi pela internet diversos posts utilizando os dados do ENEM, que são livres e abertos, disponibilizados no site do INEP. O arquivo é um pouco pesado e, por conta disso, limitarei a minha análise. O que busco aqui é entender quais variáveis e como tais variáveis afetam no desempenho em Matemática dos alunos do município do Rio de Janeiro e de São Gonçalo. As justificativas para tal escolha se dão por
- ao utilizar o Brasil inteiro, um enorme poder computacional é exigido;
- utilizando todo o país, estaríamos homogeneizando realidades distantes e assim atribuindo viés à análise. O Estado de São Paulo tem a maioria dos alunos no ENEM, e portanto enviesaria e obfuscaria aspectos que são importantes, talvez, para alunos do interior do Rio Grande do Sul;
- A cidade do Rio, além de capital, tem o segundo melhor IDH do Estado, enquanto São Gonçalo tem um dos piores, sendo duas análises distintas interessantes.
Mas como uma árvore de decisão funciona e calcula as divisões? Bom, o algoritmo calcula a média de cada preditor agrupando suas possibilidades de maneira ótima, gerando cada divisão e testando qual o melhor preditor para uma árvore, avançando na mesma em profundidade (depth), sendo que ao final do processo, valores ou categorias são apontados em cada região ou “folha”. A ideia é um pouco complexa, mas uma vez na prática, fica mais fácil de entender. Vamos para o R:
O primeiro passo é carregar os pacotes que iremos utilizar, abrir o banco e determinar uma seed, que nada mais é que um número que permite a reproducibilidade dos valores randômicos obtidos em uma pesquisa. Com a mesma seed, os mesmo valores devem ser obtidos em todos os computadores e sistemas.
# Setando seed como 93
set.seed(93)
# Carregando os pacotes
pacman::p_load(
tidyverse,
randomForest,
gbm,
tree,
fst,
ranger,
rpart,
rpart.plot,
rattle
)##
## The downloaded binary packages are in
## /var/folders/qy/4rvcxmfj4bz5vqk9hv_q9pmh0000gn/T//Rtmp1MmSXY/downloaded_packages##
## gbm installed##
## The downloaded binary packages are in
## /var/folders/qy/4rvcxmfj4bz5vqk9hv_q9pmh0000gn/T//Rtmp1MmSXY/downloaded_packages##
## tree installed# Abrindo o banco
enem2018 <- read.fst("enem2018_full.fst")Como sempre, utilizarei o tidyverse para pequenas manipulações no banco. o RandomForest, o GBM, o Tree, o Ranger, o Rpart, o Rpart.Plot e o Rattle são pacotes que utilizarei para a análise em si e lidam com árvores de decisão, florestas randômicas, bagging, boosting, e plotagem das mesmas. Já FST é um pacote que se refere ao formato .fst, que permite uma maior compressão dos dados. O banco do ENEM, completo, pesa em torno de 1.8gb em CSV, mas apenas ~800mb em FST com 100% de compressão. Em tempos de SSDs limitados, todo espaço que pode ser salvo é importante.
No arquivo baixado pelo site do INEP, temos o dicionário das variáveis, que indica o que cada variável significa. Essas devem ser trabalhadas, filtradas, alteradas. Como quero utilizar a nota de matemática, todas as outras notas devem ser removidas, e irei remover todos os casos de alunos treineiros, que ainda não tem idade/formação para entrar na faculdade e fazem a prova como um teste. Alterarei algumas variáveis também, para facilitar a leitura das mesmas.
enem2018 <- enem2018 %>%
# Filtrando só quem não é treineiro e que participou da prova de matemática
filter(IN_TREINEIRO == 0,
TP_PRESENCA_MT == 1) %>%
# Removendo as variáveis que não utilizarei, mantendo somente as que podem se relacionar com a nota, como as variáveis do questionário socioeconômico, representadas pela inicial "Q".
dplyr::select(
-c(
starts_with("TX"),
starts_with("CO"),
starts_with("IN"),
starts_with("NU_NOTA_COMP"),
starts_with("TP_P"),
NU_ANO,
NU_INSCRICAO,
starts_with("SG_UF_R"),
TP_NACIONALIDADE,
TP_ESTADO_CIVIL,
TP_ST_CONCLUSAO,
TP_ANO_CONCLUIU,
TP_ENSINO,
TP_SIT_FUNC_ESC,
NU_NOTA_CN,
NU_NOTA_CH,
NU_NOTA_LC,
TP_LINGUA,
TP_STATUS_REDACAO,
NU_NOTA_REDACAO,
SG_UF_NASCIMENTO
)
) %>%
# Como acredito que o tipo de escola possa vir a influenciar na variável dependente da nota,
# vou alterar para aparecer cada caso,assim como com a raça e a localização.
mutate(
TP_ESCOLA = case_when(
TP_ESCOLA == 1 ~ "NR",
TP_ESCOLA == 2 ~ "Pública",
TP_ESCOLA == 3 ~ "Privada",
TP_ESCOLA == 4 ~ "Exterior"
),
TP_DEPENDENCIA_ADM_ESC = case_when(
TP_DEPENDENCIA_ADM_ESC == 1 ~ "Federal",
TP_DEPENDENCIA_ADM_ESC == 2 ~ "Estadual",
TP_DEPENDENCIA_ADM_ESC == 3 ~ "Municipal",
TP_DEPENDENCIA_ADM_ESC == 4 ~ "Privada"
),
TP_LOCALIZACAO_ESC = case_when(
TP_LOCALIZACAO_ESC == 1 ~ "Urbana",
TP_LOCALIZACAO_ESC == 2 ~ "Rural"
),
TP_COR_RACA = case_when(
TP_COR_RACA == 0 ~ "Não declarado",
TP_COR_RACA == 1 ~ "Branca",
TP_COR_RACA == 2 ~ "Preta",
TP_COR_RACA == 3 ~ "Parda",
TP_COR_RACA == 4 ~ "Amarela",
TP_COR_RACA == 5 ~ "Indígena"
)
) %>%
mutate_if(is.character, as.factor) %>%
mutate_at(vars(starts_with("TP")), as.factor) %>%
mutate(Q005 = as.factor(Q005))O próximo passo seria filtrar os alunos cuja escola é no município do Rio e em São Gonçalo, criando dois objetos para tais. Excluirei também os alunos que afirmam terem estudado no Exterior e os que não responderam.
enem2018rio <- enem2018 %>%
filter(
SG_UF_ESC == "RJ",
NO_MUNICIPIO_ESC == "Rio de Janeiro",
TP_ESCOLA != "Exterior",
TP_ESCOLA != "NR"
) %>%
select(-c(starts_with("NO_MUN"),
starts_with("SG_UF")))
enem2018sg <- enem2018 %>%
filter(
SG_UF_ESC == "RJ",
NO_MUNICIPIO_ESC == "São Gonçalo",
TP_ESCOLA != "Exterior",
TP_ESCOLA != "NR"
) %>%
select(-c(starts_with("NO_MUN"),
starts_with("SG_UF")))Usando o Tree
Pronto, a partir de agora podemos criar as nossas primeiras árvores de decisão para comparar o Rio e São Gonçalo. As árvores são criadas pelo comando tree(), que obedecem à mesma sintaxe de um lm(), com “variável dependente ~ preditores, data”.
tree.enemrio <- tree(NU_NOTA_MT ~., data = enem2018rio)
tree.enemsg <- tree(NU_NOTA_MT~., data = enem2018sg)
summary(tree.enemrio)##
## Regression tree:
## tree(formula = NU_NOTA_MT ~ ., data = enem2018rio)
## Variables actually used in tree construction:
## [1] "TP_SEXO" "Q006"
## Number of terminal nodes: 4
## Residual mean deviance: 6680 = 102000000 / 15200
## Distribution of residuals:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -573.00 -61.00 -8.69 0.00 53.10 374.00summary(tree.enemsg)##
## Regression tree:
## tree(formula = NU_NOTA_MT ~ ., data = enem2018sg)
## Variables actually used in tree construction:
## [1] "TP_SEXO" "NU_IDADE"
## Number of terminal nodes: 3
## Residual mean deviance: 5660 = 14600000 / 2580
## Distribution of residuals:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -149.00 -58.20 -9.07 0.00 49.00 263.00Com o comando summary() dado nas árvores acima, temos os preditores utilizados pelo algoritmo para dividir os casos: TP_SEXO e Q006, no Rio, e TP_SEXO e NU_IDADE em São Gonçalo. A primeira variável se refere ao gênero do aluno, enquanto a Q006 se refere à renda famíliar do aluno. A variável NU_IDADE é, como se pressupõe, a idade do aluno. O summary() nos dá outras informações interessantes, e a principal delas por agora é o número de nós terminais: 4 e 3, respectivamente. Podemos plotar essas árvores para compreendê-las melhor:
par(mfrow=c(1,2))
plot(tree.enemrio)
text(tree.enemrio)
title(main = "Rio de Janeiro")
plot(tree.enemsg)
text(tree.enemsg)
title(main = "São Gonçalo")
Como podemos interpretar esses dois gráficos?
No caso do Rio, se o aluno for do sexo feminino e estiver em uma família cuja renda mensal vai de zero até R$ 1908,00, a nota em matemática gira em torno da média de 493.8. Se for mulher mas tiver renda superior a R$1908,01 , ou seja, mais abastada em comparação ao outro grupo, a nota de matemática tende a aumentar, ficando em torno da média de 525. Agora, quando se é homem, o que é representado pelo lado direito do primeiro plot, mas com renda até R$ 2385,00, a nota ainda se posiciona perto de 532.8, enquanto as rendas superiores a tal valor recebem a nota de 572.6.
Em São Gonçalo, a renda não influencia a ponto de aparecer no gráfico, mas sim o sexo e a idade. Mulheres com menos de 18,5 anos recebem a nota, em média, de 500,1, enquanto as mais velhas giram em torno de 471,9. Todavia, a idade não influencia na nota dos homens, que se localiza ao redor de 522,9.
Tivemos sorte que as nossas duas árvores são simples. Algumas árvores podem ser absurdamente complexas, com alta profundidade, utilizando vários preditores e tendo vários nós terminais, o que dificulta a interpretação. Para isso, podemos utilizar o prune, que nada mais é que reduzir ou “podar” nossas árvores para o ponto com o menor deviance. Para fazer isso, fazemos uma validação cruzada da ávore, buscando identificar o número de nodes que leva ao menor desvio. Vamos verificar isso com o comando cv.tree():
cv.rio <- cv.tree(tree.enemrio, K = 100)
cv.sg <- cv.tree(tree.enemsg, K = 100)
par(mfrow=c(1,2))
plot(cv.rio$size, cv.rio$dev, type = "b")
title(main = "Rio de Janeiro")
plot(cv.sg$size, cv.sg$dev, type = "b")
title(main = "São Gonçalo")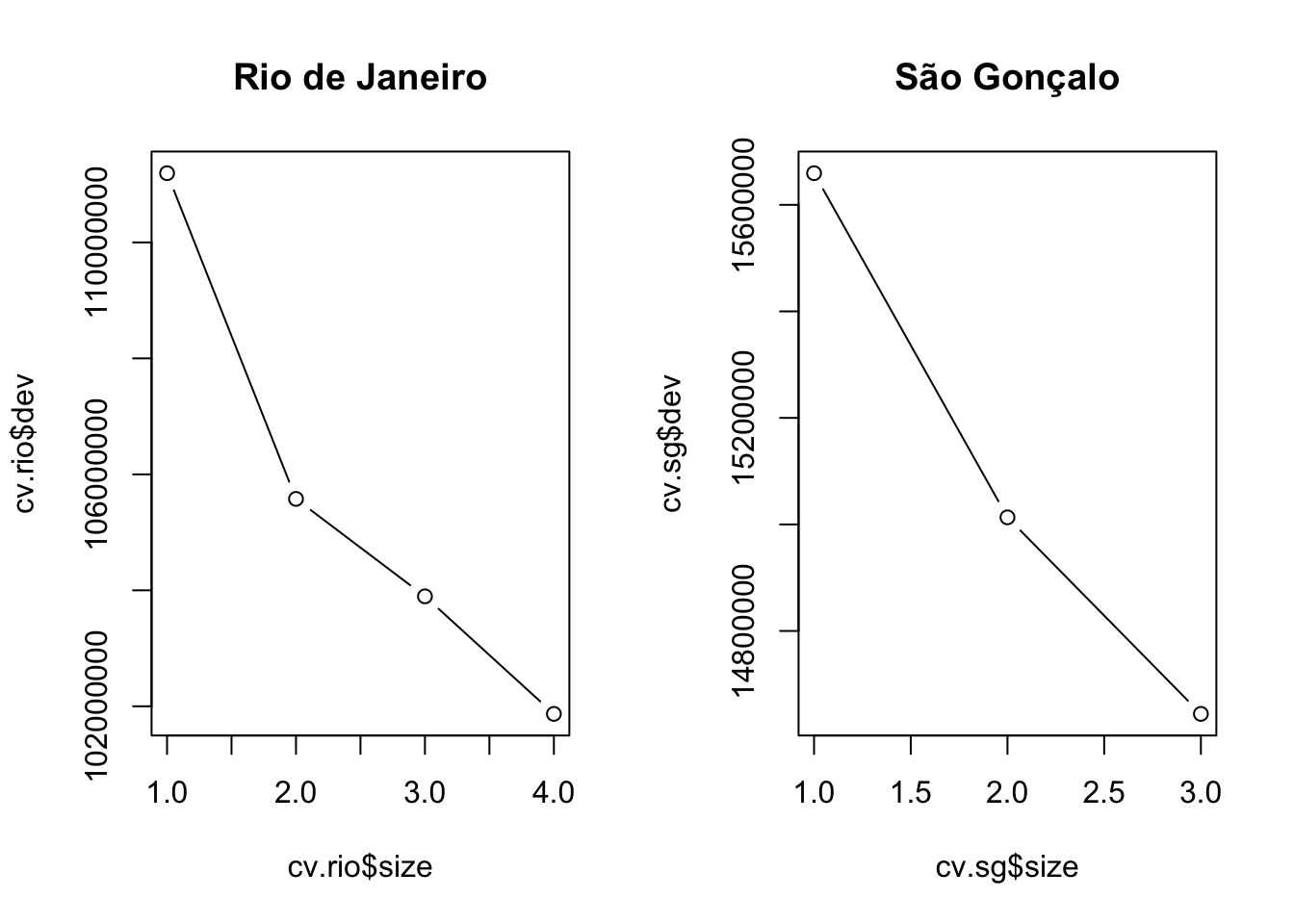
Como podemos ver, o menor valor é encontrado quando a árvore possui respectivamente 4 e 3 nós terminais, ou seja, já temos a árvore com o menor deviance. Mas se quiséssemos fazer o prune, utilizaríamos o seguinte comando:
prune.enem <- prune.tree(tree.enemrio, best = 4)No argumento best, colocamos o número de nós terminais que desejamos, de acordo com a validação cruzada feita anteriormente.
Usando o rPart
Podemos também fazer as mesmas árvores usando o pacote rPart, que em conjunto com o rpart.plot e o rattle, nos permite a plotagem de árvores com melhor apelo visual. A sintaxe é basicamente a mesma:
rpart.rio <- rpart(NU_NOTA_MT ~ ., data = enem2018rio)
rpart.sg <- rpart(NU_NOTA_MT ~ ., data = enem2018sg)
# No Rio
summary(rpart.rio)## Call:
## rpart(formula = NU_NOTA_MT ~ ., data = enem2018rio)
## n= 15233
##
## CP nsplit rel error xerror xstd
## 1 0.0518975 0 1.000000 1.000092 0.0117093
## 2 0.0158478 1 0.948103 0.948297 0.0110942
## 3 0.0152261 2 0.932255 0.936489 0.0109793
## 4 0.0100000 3 0.917029 0.918746 0.0106895
##
## Variable importance
## TP_SEXO Q006 Q024 Q002 Q003 Q001 Q010
## 58 35 2 2 1 1 1
##
## Node number 1: 15233 observations, complexity param=0.0518975
## mean=517.931, MSE=7287.81
## left son=2 (9198 obs) right son=3 (6035 obs)
## Primary splits:
## TP_SEXO splits as LR, improve=0.0518975, (0 missing)
## Q006 splits as LLLLLRRRRRRRRRRRR, improve=0.0378755, (0 missing)
## Q024 splits as LRRRR, improve=0.0196952, (0 missing)
## NU_IDADE < 18.5 to the right, improve=0.0193174, (0 missing)
## Q003 splits as LLLRRL, improve=0.0192026, (0 missing)
## Surrogate splits:
## Q006 splits as LLLLLLLRLLRRLRRRR, agree=0.607, adj=0.007, (0 split)
## Q024 splits as LLRRR, agree=0.606, adj=0.005, (0 split)
## Q014 splits as LLRLR, agree=0.605, adj=0.003, (0 split)
## Q005 splits as RLLLLLLLLLLLRL-LRL-L, agree=0.605, adj=0.002, (0 split)
## Q016 splits as LLR-R, agree=0.605, adj=0.002, (0 split)
##
## Node number 2: 9198 observations, complexity param=0.0158478
## mean=502.178, MSE=5880.98
## left son=4 (6729 obs) right son=5 (2469 obs)
## Primary splits:
## Q006 splits as LLLLRRRRRRRRRRRRR, improve=0.0325244, (0 missing)
## NU_IDADE < 18.5 to the right, improve=0.0225858, (0 missing)
## Q001 splits as LLLLRRRL, improve=0.0179838, (0 missing)
## Q002 splits as LLLLRRRL, improve=0.0168509, (0 missing)
## Q003 splits as LLLRRL, improve=0.0162138, (0 missing)
## Surrogate splits:
## Q002 splits as LLLLLRRL, agree=0.748, adj=0.061, (0 split)
## Q024 splits as LLRRR, agree=0.745, adj=0.050, (0 split)
## Q001 splits as LLLLLRRL, agree=0.741, adj=0.036, (0 split)
## Q003 splits as LLLRRL, agree=0.739, adj=0.029, (0 split)
## Q010 splits as LLRRR, agree=0.737, adj=0.019, (0 split)
##
## Node number 3: 6035 observations, complexity param=0.0152261
## mean=541.941, MSE=8477.31
## left son=6 (4647 obs) right son=7 (1388 obs)
## Primary splits:
## Q006 splits as LLLLLRRRRRRRRRRRR, improve=0.0330397, (0 missing)
## NU_IDADE < 18.5 to the right, improve=0.0260023, (0 missing)
## Q003 splits as LLLRRL, improve=0.0191681, (0 missing)
## Q002 splits as LLLLRRRL, improve=0.0188350, (0 missing)
## Q001 splits as LLLLRRRL, improve=0.0180970, (0 missing)
## Surrogate splits:
## Q002 splits as LLLLLLRL, agree=0.780, adj=0.043, (0 split)
## Q024 splits as LLLRR, agree=0.779, adj=0.038, (0 split)
## Q010 splits as LLRRL, agree=0.777, adj=0.031, (0 split)
## Q003 splits as LLLLRL, agree=0.777, adj=0.030, (0 split)
## Q001 splits as LLLLLLRL, agree=0.775, adj=0.021, (0 split)
##
## Node number 4: 6729 observations
## mean=493.801, MSE=5212.72
##
## Node number 5: 2469 observations
## mean=525.01, MSE=6989.69
##
## Node number 6: 4647 observations
## mean=532.794, MSE=7611.91
##
## Node number 7: 1388 observations
## mean=572.563, MSE=10156.9# Em São Gonçalo
summary(rpart.sg)## Call:
## rpart(formula = NU_NOTA_MT ~ ., data = enem2018sg)
## n= 2583
##
## CP nsplit rel error xerror xstd
## 1 0.0354531 0 1.000000 1.000863 0.0277142
## 2 0.0158005 1 0.964547 0.966190 0.0264783
## 3 0.0100000 2 0.948746 0.950902 0.0259432
##
## Variable importance
## TP_SEXO NU_IDADE Q005 Q024 Q002
## 67 30 1 1 1
##
## Node number 1: 2583 observations, complexity param=0.0354531
## mean=504.133, MSE=5955.64
## left son=2 (1613 obs) right son=3 (970 obs)
## Primary splits:
## TP_SEXO splits as LR, improve=0.0354531, (0 missing)
## NU_IDADE < 18.5 to the right, improve=0.0207137, (0 missing)
## Q001 splits as LLLLRRLL, improve=0.0159758, (0 missing)
## Q006 splits as LLLRRRLRRLRRLLR-L, improve=0.0153501, (0 missing)
## Q002 splits as LLLRRRRL, improve=0.0128527, (0 missing)
## Surrogate splits:
## Q024 splits as LLRRL, agree=0.630, adj=0.013, (0 split)
## Q002 splits as LLLLLLRL, agree=0.627, adj=0.006, (0 split)
## Q006 splits as LLLLLLLLLRLRLLL-L, agree=0.626, adj=0.003, (0 split)
## Q005 splits as LLLLLLLRLL----------, agree=0.625, adj=0.002, (0 split)
## Q010 splits as LLLRR, agree=0.625, adj=0.002, (0 split)
##
## Node number 2: 1613 observations, complexity param=0.0158005
## mean=492.864, MSE=5310.8
## left son=4 (412 obs) right son=5 (1201 obs)
## Primary splits:
## NU_IDADE < 18.5 to the right, improve=0.02837470, (0 missing)
## Q001 splits as LLLLRRLL, improve=0.02151170, (0 missing)
## Q006 splits as LLRRRRRRRLRRLLR-R, improve=0.01664760, (0 missing)
## Q002 splits as LLLRRRRL, improve=0.01389590, (0 missing)
## Q013 splits as LRRRL, improve=0.00986575, (0 missing)
## Surrogate splits:
## Q005 splits as LRRRRRRLRR----------, agree=0.751, adj=0.027, (0 split)
## Q002 splits as LRRRRRRR, agree=0.746, adj=0.005, (0 split)
## Q006 splits as RRRRRRRRRRRRRLL-R, agree=0.746, adj=0.005, (0 split)
## Q027 splits as RRRLR-, agree=0.745, adj=0.002, (0 split)
##
## Node number 3: 970 observations
## mean=522.871, MSE=6465.7
##
## Node number 4: 412 observations
## mean=471.906, MSE=3658.29
##
## Node number 5: 1201 observations
## mean=500.054, MSE=5675.3O summary() acima nos mostra uma informação importantíssima: a importância de cada variável dentro do conjunto utilizado. No caso do Rio, percebemos que TP_SEXO e Q006, as duas primeiras, estão muito acima das outras variáveis, como a Q024, referente a existência de computador na residência. Isso justifica a utilização das duas primeiras mas não do resto. No caso de São Gonçalo, é possível perceber que TP_SEXO é mais de duas vezes mais importante que NU_IDADE, que por sua vez é bem distante de Q005, que versa sobre a quantidade de pessoas na residência. Como são pouco importantes no cálculo final, não foram utilizadas na árvore.
Para ver as regras da árvore, temos o comando rpart.rules(), que é usado da seguinte forma:
rpart.rules(rpart.rio)## NU_NOTA_MT
## 494 when TP_SEXO is F & Q006 is A or B or C or D
## 525 when TP_SEXO is F & Q006 is E or F or G or H or I or J or K or L or M or N or O or P or Q
## 533 when TP_SEXO is M & Q006 is A or B or C or D or E
## 573 when TP_SEXO is M & Q006 is F or G or H or I or J or K or L or M or N or O or P or Qrpart.rules(rpart.sg)## NU_NOTA_MT
## 472 when TP_SEXO is F & NU_IDADE >= 19
## 500 when TP_SEXO is F & NU_IDADE < 19
## 523 when TP_SEXO is ME para a plotagem das árvores, utilizo o fancyRpartPlot():
# Para o Rio
rattle::fancyRpartPlot(rpart.rio, sub = NULL)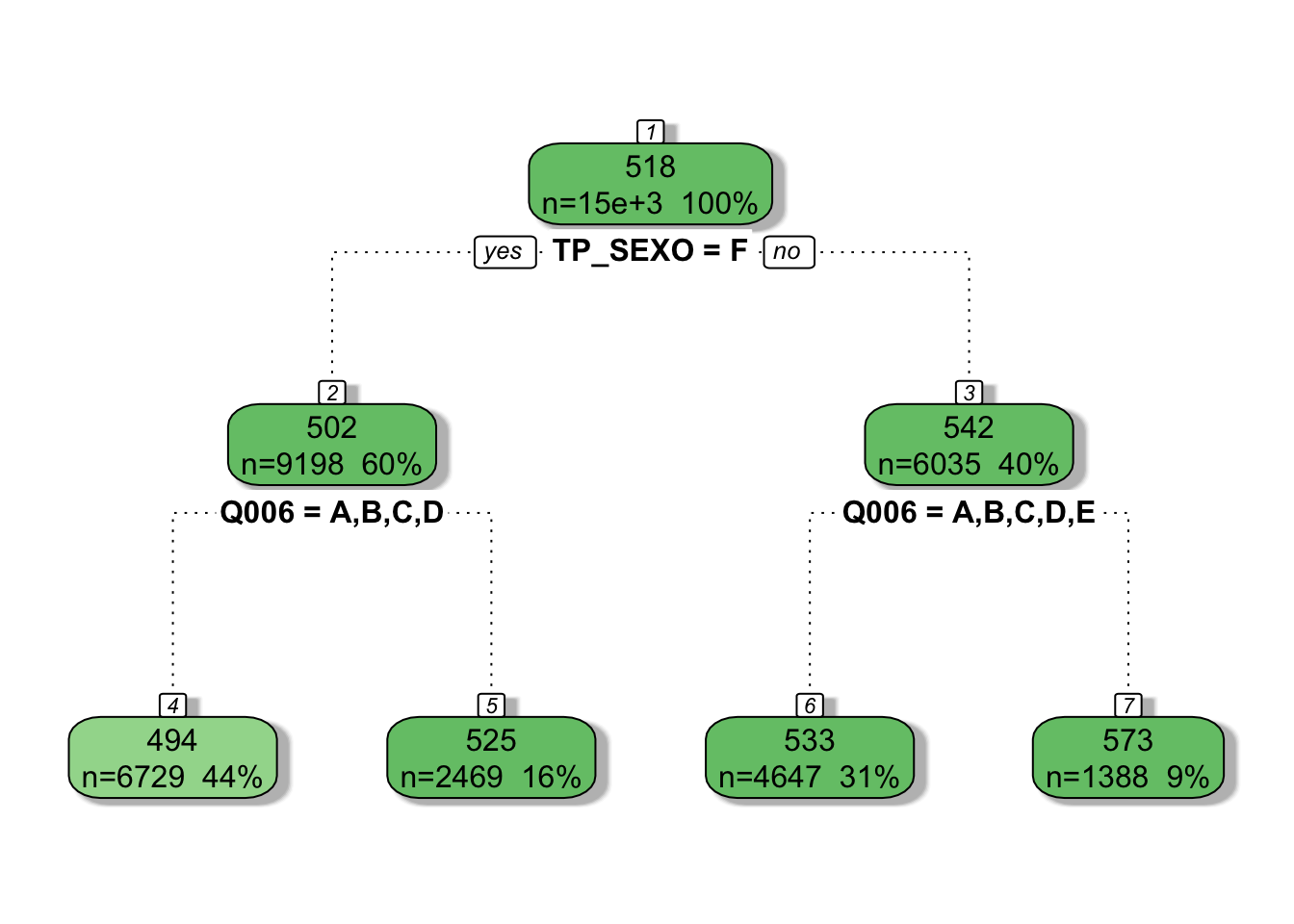
# Para São Gonçalo
rattle::fancyRpartPlot(rpart.sg, sub = NULL)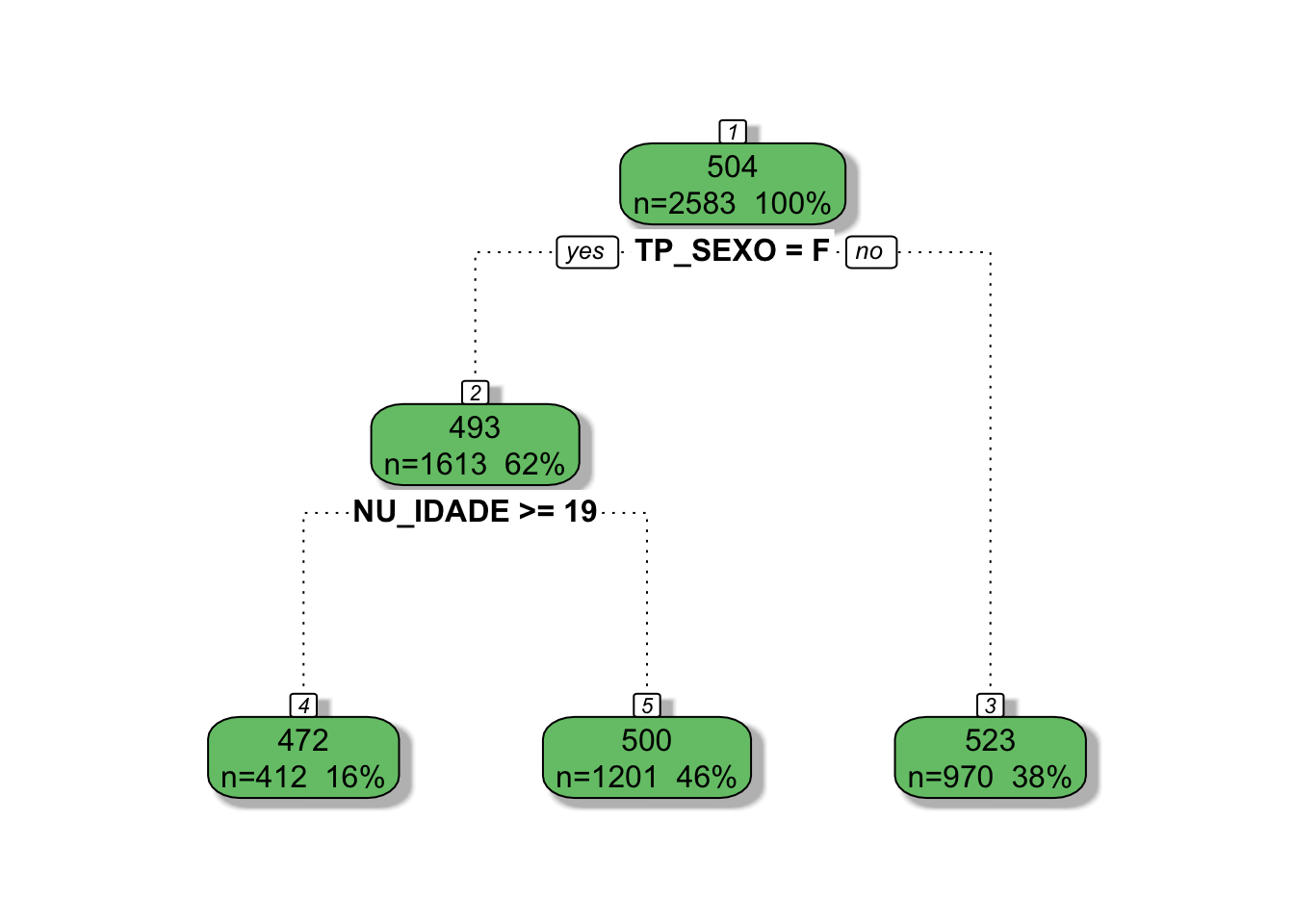
Uma vantagem do rPart é que alguns aspectos no cálculo podem ser alterados. Poderíamos tornar a árvore mais complexa alterando o complexity parameter, o que poderia aumentar o número de nós, preditores e nós terminais. Para isso, bastaria aperfeiçoar o comando da seguinte maneira:
rpart.rio2 <- rpart(NU_NOTA_MT ~ ., enem2018rio, control = rpart.control(cp = .003))
rpart.sg2 <- rpart(NU_NOTA_MT ~ ., enem2018sg, control = rpart.control(cp = .005))
fancyRpartPlot(rpart.rio2, sub = NULL)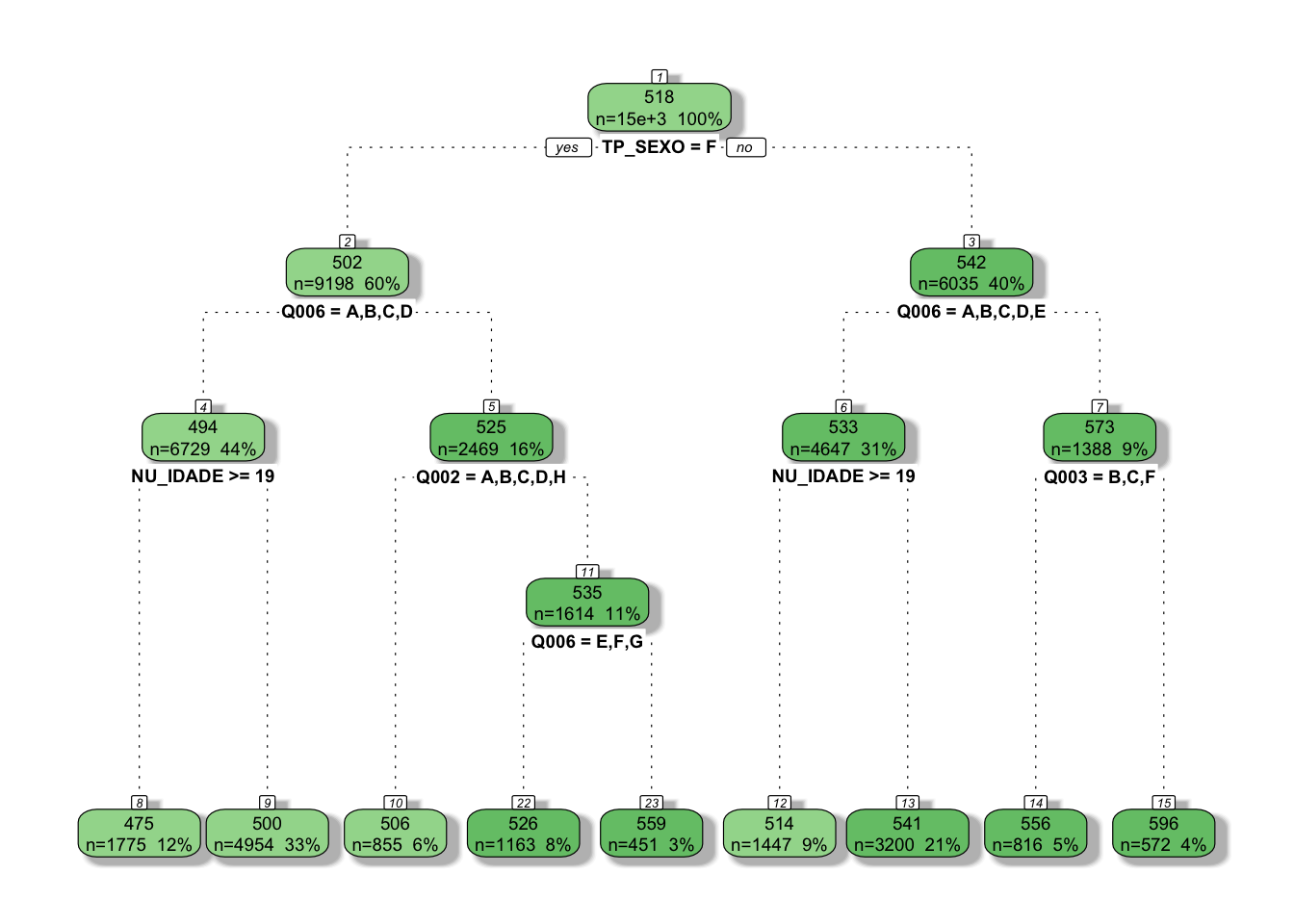
fancyRpartPlot(rpart.sg2, sub = NULL)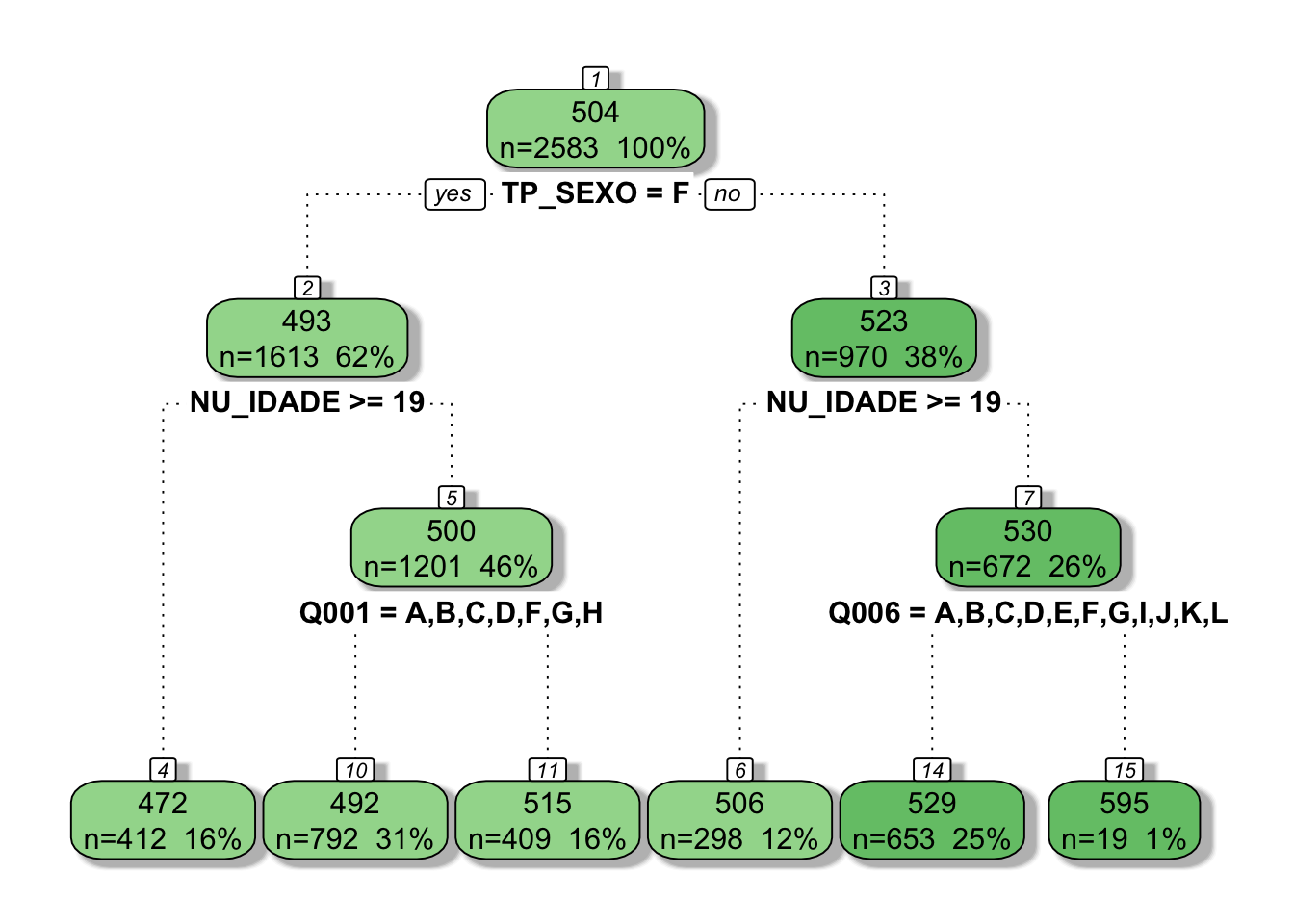
Se compararmos o erro relativo nas duas árvores em cada cidade, percebemos que o mesmo se altera quando a complexidade é maior. Todavia, isso também dificulta a interpretação da mesma e adiciona viés.
par(mfrow=c(2,2))
plotcp(rpart.rio)
plotcp(rpart.rio2)
plotcp(rpart.sg)
plotcp(rpart.sg)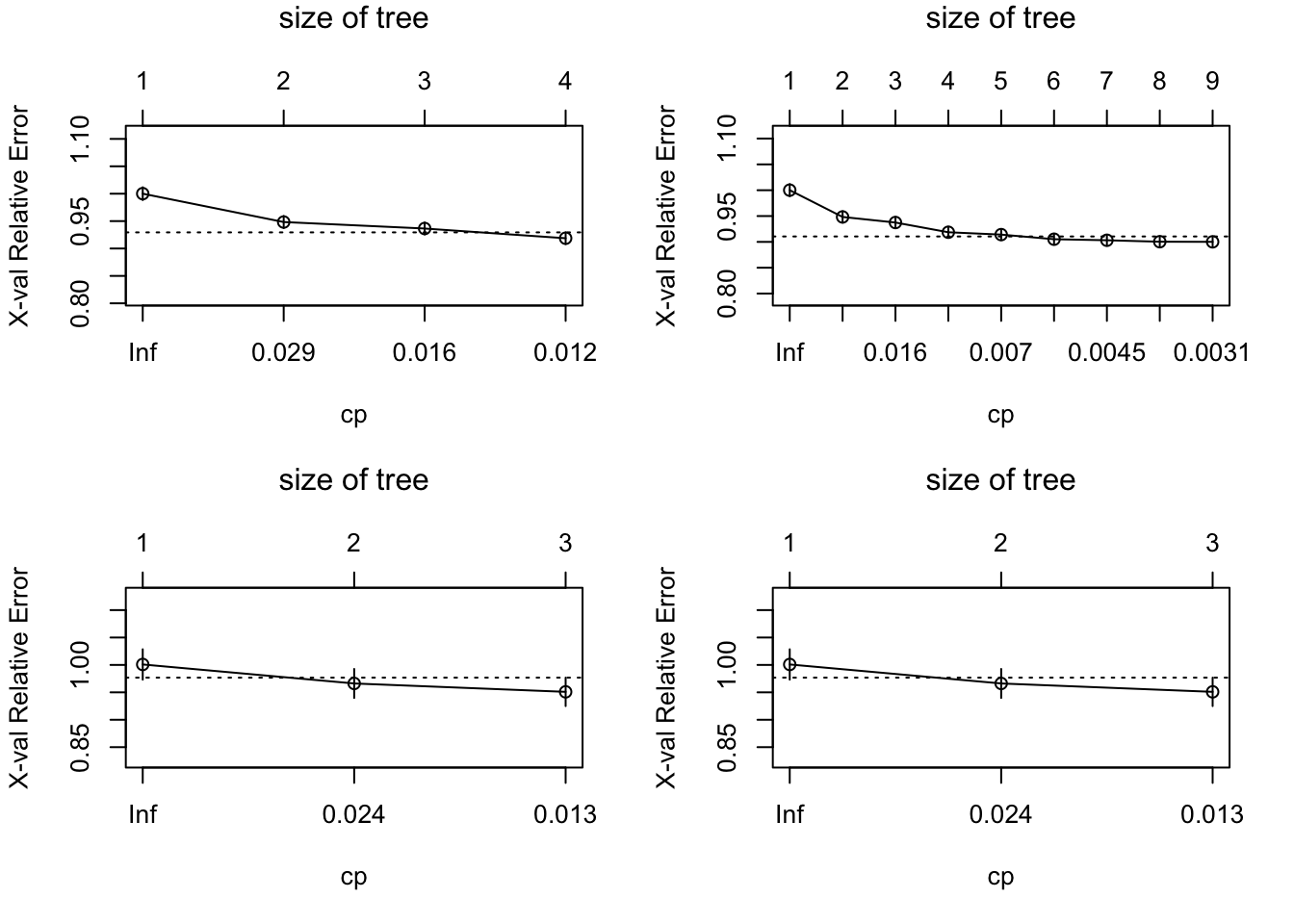
No caso do Rio, o erro relativo diminui, enquanto o menor erro em São Gonçalo é quando a árvore tem apenas 3 nós finais, aumentando suavemente depois.
Bagging, RandomForest e Boosting
Entretanto, quando fazemos essa árvore, que garantia temos de que essa é a melhor árvore possível? Testando com técnicas de bootstrapping, repetindo a criação de árvores centenas ou milhares de vezes, buscando a média dos resultados para ver como a árvore se adapta e quais os melhores preditores usados, é claro!
Bagging
A primeira técnica que podemos utilizar é de bagging, que faz uma amostragem de casos usando todos os preditores. Para fazer o bagging, o pacote randomForest é fundamental. Todavia, quando lidamos com bancos muito grandes, é mais interessante usar o ranger, que permite a utilização de todos os núcleos do processador presentes na máquina.
bag.enemrio <- ranger(
NU_NOTA_MT ~ .,
data = enem2018rio,
mtry = 33,
importance = "impurity",
num.threads = 12,
num.trees = 1000
)
bag.enemsg <- ranger(
NU_NOTA_MT ~ .,
data = enem2018sg,
mtry = 33,
importance = "impurity",
num.threads = 12,
num.trees = 1000
)Acima, criamos 1000 árvores utilizando os 33 preditores presentes no banco (o 34º é NU_NOTA_MT, claro)
bag.enemrio## Ranger result
##
## Call:
## ranger(NU_NOTA_MT ~ ., data = enem2018rio, mtry = 33, importance = "impurity", num.threads = 12, num.trees = 1000)
##
## Type: Regression
## Number of trees: 1000
## Sample size: 15233
## Number of independent variables: 33
## Mtry: 33
## Target node size: 5
## Variable importance mode: impurity
## Splitrule: variance
## OOB prediction error (MSE): 6471.47
## R squared (OOB): 0.112074bag.enemsg## Ranger result
##
## Call:
## ranger(NU_NOTA_MT ~ ., data = enem2018sg, mtry = 33, importance = "impurity", num.threads = 12, num.trees = 1000)
##
## Type: Regression
## Number of trees: 1000
## Sample size: 2583
## Number of independent variables: 33
## Mtry: 33
## Target node size: 5
## Variable importance mode: impurity
## Splitrule: variance
## OOB prediction error (MSE): 5802.14
## R squared (OOB): 0.0261514O \(R^{2}_{OOB}\) presente ao final de cada um dos comandos é a capacidade explicativa daquela árvore para aquele conjunto de dados. Uma informação interessante e importante que, de certa forma, aprimora as nossas primeiras árvores, é a importância das variáveis, que pode ser buscada em:
bag.enemrio$variable.importance## NU_IDADE TP_SEXO TP_COR_RACA
## 6378165.60 5754041.08 4714652.67
## TP_ESCOLA TP_DEPENDENCIA_ADM_ESC TP_LOCALIZACAO_ESC
## 100129.80 107150.24 0.00
## Q001 Q002 Q003
## 6943347.67 6794857.41 5580028.69
## Q004 Q005 Q006
## 4725148.02 7188983.55 9422883.20
## Q007 Q008 Q009
## 1042060.45 2357135.74 3892465.06
## Q010 Q011 Q012
## 2519658.59 1019978.23 1048116.99
## Q013 Q014 Q015
## 2878266.93 1934756.28 1353276.84
## Q016 Q017 Q018
## 2277991.45 355740.36 1770814.11
## Q019 Q020 Q021
## 3687053.88 2318066.98 2251439.30
## Q022 Q023 Q024
## 5108939.28 2361920.88 3689504.05
## Q025 Q026 Q027
## 1478319.86 5746.98 2740928.04bag.enemsg$variable.importance## NU_IDADE TP_SEXO TP_COR_RACA
## 965826.102 552091.844 755000.653
## TP_ESCOLA TP_DEPENDENCIA_ADM_ESC TP_LOCALIZACAO_ESC
## 355.724 16998.611 16013.748
## Q001 Q002 Q003
## 967023.439 995490.596 790064.877
## Q004 Q005 Q006
## 668391.870 943918.888 1144531.549
## Q007 Q008 Q009
## 104452.466 305429.915 567387.939
## Q010 Q011 Q012
## 345626.179 125020.846 145582.724
## Q013 Q014 Q015
## 373738.937 351303.316 206192.739
## Q016 Q017 Q018
## 330894.741 66615.938 185943.435
## Q019 Q020 Q021
## 509809.443 334796.907 287964.339
## Q022 Q023 Q024
## 794836.643 328621.680 498336.834
## Q025 Q026 Q027
## 225205.884 15175.975 409283.488Para vermos as 6 variáveis mais importantes de cada banco, basta utilizarmos os seguintes comandos:
# Para o Rio
bag.enemrio$variable.importance %>%
as.data.frame() %>%
rename("imp" = 1) %>%
rownames_to_column() %>%
arrange(-imp) %>%
head(6)## rowname imp
## 1 Q006 9422883
## 2 Q005 7188984
## 3 Q001 6943348
## 4 Q002 6794857
## 5 NU_IDADE 6378166
## 6 TP_SEXO 5754041# Para São Gonçalo
bag.enemsg$variable.importance %>%
as.data.frame() %>%
rename("imp" = 1) %>%
rownames_to_column() %>%
arrange(-imp) %>%
head(6)## rowname imp
## 1 Q006 1144532
## 2 Q002 995491
## 3 Q001 967023
## 4 NU_IDADE 965826
## 5 Q005 943919
## 6 Q022 794837As variáveis novas que ali surgiram e não foram citadas anteriormente são:
- Q005 - número de pessoas que moram com o aluno;
- Q001 - até que série o pai estudou;
- Q002 - até que série a mãe estudou
- Q003 - a ocupação do pai
RandomForests
O RandomForests nada mais é que o sampling criado pelo bagging, porém utilizando apenas um determinado número de preditores, e não o máximo. Quando colocamos todos os preditores, todos eles estarão em todas as árvores. É possível que algum seja obfuscado por outro, pouco mais forte, o que impede que o mesmo seja visível. Com apenas um subsetting dos preditores, podemos fazer um “rodízio” das variáveis, não colocando todas ao mesmo tempo e analisando como elas, então, se comportam. O comando é o mesmo, mudando apenas o argumento mtry, que indica o número de preditores usados.
rf.enemrio <- ranger(
NU_NOTA_MT ~ .,
data = enem2018rio,
mtry = 11,
importance = "impurity",
num.threads = 12,
verbose = T,
num.trees = 1000,
)
rf.enemsg <- ranger(
NU_NOTA_MT ~ .,
data = enem2018sg,
mtry = 11,
importance = "impurity",
num.threads = 12,
verbose = T,
num.trees = 1000,
)Coloquei 11 preditores para que \(\frac{1}{3}\) do total seja utilizado e possamos diminuir o viés descorrelacionando as árvores, o que não acontece no bagging.
rf.enemrio## Ranger result
##
## Call:
## ranger(NU_NOTA_MT ~ ., data = enem2018rio, mtry = 11, importance = "impurity", num.threads = 12, verbose = T, num.trees = 1000, )
##
## Type: Regression
## Number of trees: 1000
## Sample size: 15233
## Number of independent variables: 33
## Mtry: 11
## Target node size: 5
## Variable importance mode: impurity
## Splitrule: variance
## OOB prediction error (MSE): 6354.81
## R squared (OOB): 0.128079rf.enemsg## Ranger result
##
## Call:
## ranger(NU_NOTA_MT ~ ., data = enem2018sg, mtry = 11, importance = "impurity", num.threads = 12, verbose = T, num.trees = 1000, )
##
## Type: Regression
## Number of trees: 1000
## Sample size: 2583
## Number of independent variables: 33
## Mtry: 11
## Target node size: 5
## Variable importance mode: impurity
## Splitrule: variance
## OOB prediction error (MSE): 5685.47
## R squared (OOB): 0.0457337Notamos um aumento no \(R^{2}_{OOB}\) em ambos, ou seja, aprimoramos o modelo. E a importância das variáveis?
# Para o Rio
rf.enemrio$variable.importance %>%
as.data.frame() %>%
rename("imp" = 1) %>%
rownames_to_column() %>%
arrange(-imp) %>%
head(6)## rowname imp
## 1 Q006 9193883
## 2 Q005 6647643
## 3 Q001 6344146
## 4 NU_IDADE 6269901
## 5 Q002 6124216
## 6 TP_SEXO 5355912# Para São Gonçalo
rf.enemsg$variable.importance %>%
as.data.frame() %>%
rename("imp" = 1) %>%
rownames_to_column() %>%
arrange(-imp) %>%
head(6)## rowname imp
## 1 Q006 1039199
## 2 Q002 918182
## 3 NU_IDADE 914388
## 4 Q001 909747
## 5 Q005 863187
## 6 Q022 752462Comparando com os resultados anteriores, percebemos apenas uma mudança da ordem de importância no Rio,e a troca da variável Q003 para Q022 em São Gonçalo. Essa variável questiona sobre a existência de telefone celular na residência.
Percebemos uma melhora no RandomForests em relação ao bagging! Mas ainda há uma abordagem para vermos, o boosting.
Boosting
Enquanto o bagging e o randomforest fazem bootstrapping, ou seja, calculam centenas ou milhares de árvores e depois unem esse resultado para uma média, o boosting cria várias árvores baseadas no resíduo da árvore anterior, ou seja, a árvore cresce sequencialmente, sendo aprimorada nas partes com maior erro no modelo anterior. O pacote que permite o boosting é o GBM, onde conseguimos limitar o máximo da profundidade possível:
boost.enemrio <-
gbm(NU_NOTA_MT ~ ., data = enem2018rio, distribution = "gaussian",
n.trees = 5000, interaction.depth = 6, n.cores = 12)
boost.enemsg <-
gbm(NU_NOTA_MT ~ ., data = enem2018sg, distribution = "gaussian",
n.trees = 5000, interaction.depth = 6, n.cores = 12)Ao fazermos o sumário dos dados, temos a importância de cada variável, em ordem. Observando as 9 primeiras de cada um:
summary(boost.enemrio) %>%
head(9)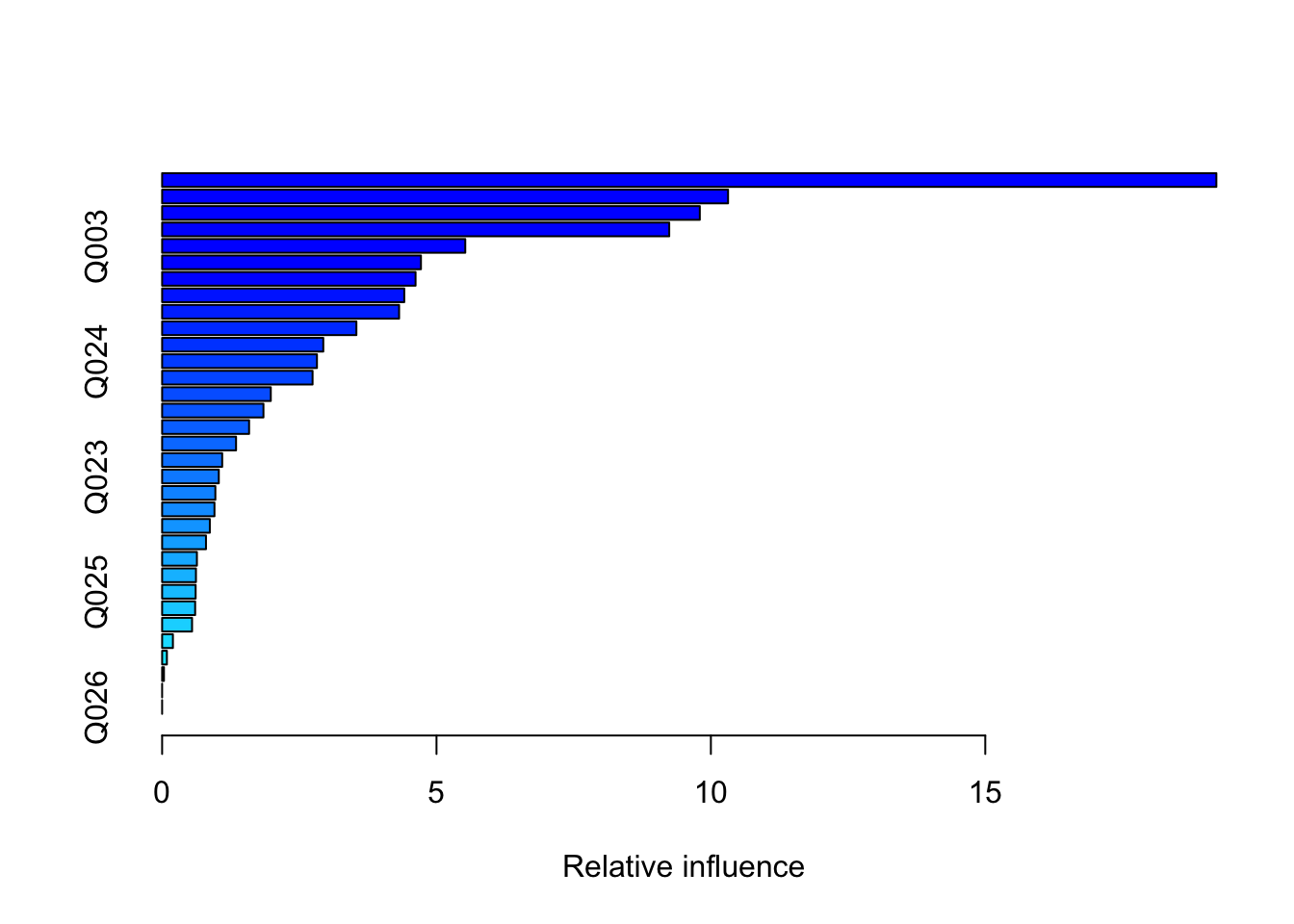
## var rel.inf
## Q006 Q006 19.21134
## Q001 Q001 10.31323
## Q005 Q005 9.79820
## Q002 Q002 9.24116
## Q003 Q003 5.52420
## Q004 Q004 4.71671
## Q022 Q022 4.61842
## NU_IDADE NU_IDADE 4.41251
## TP_COR_RACA TP_COR_RACA 4.31856summary(boost.enemsg) %>%
head(9)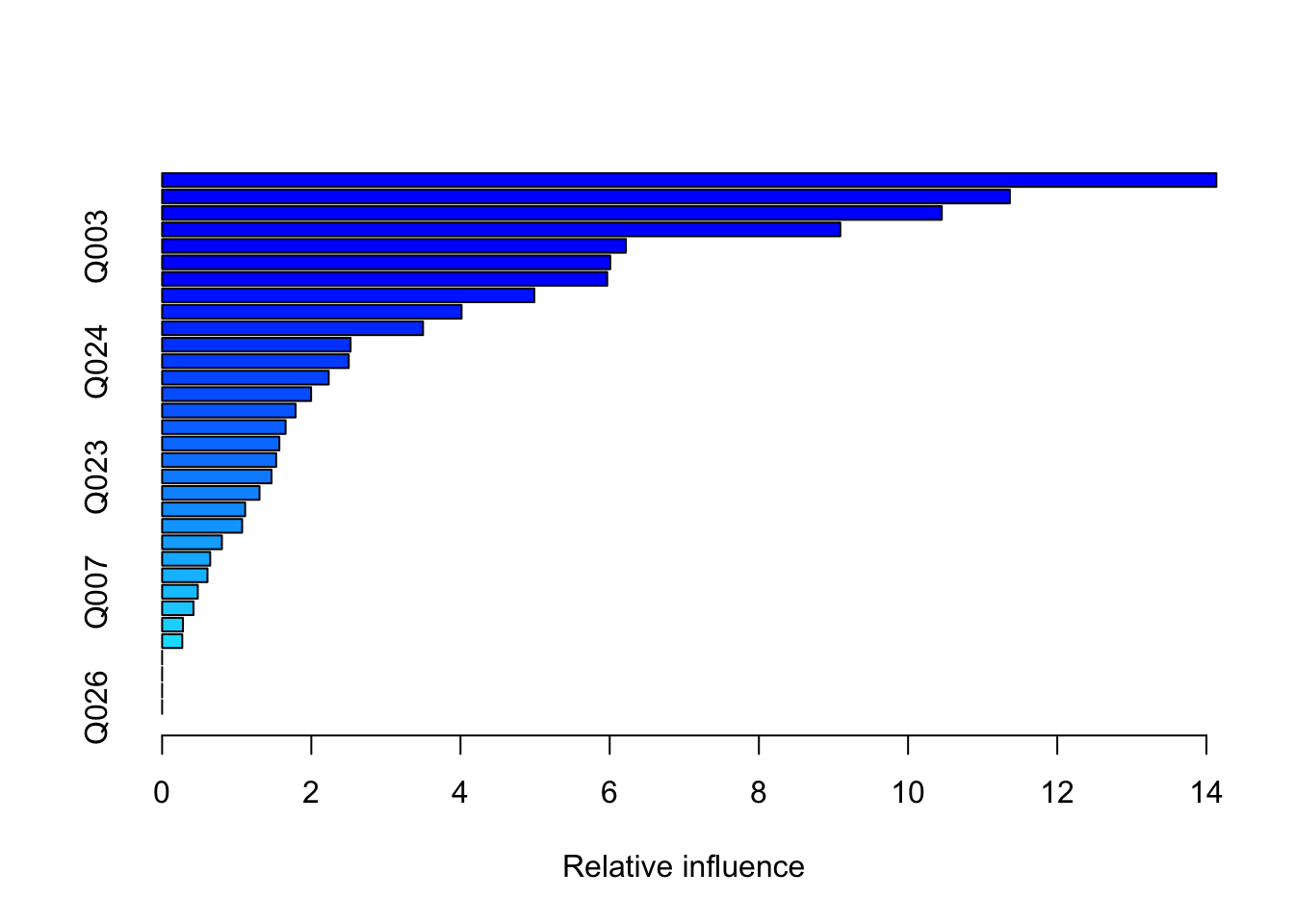
## var rel.inf
## Q006 Q006 14.13277
## Q001 Q001 11.36461
## Q002 Q002 10.45213
## Q005 Q005 9.09167
## Q003 Q003 6.21679
## Q022 Q022 6.00759
## TP_COR_RACA TP_COR_RACA 5.96695
## Q004 Q004 4.99026
## NU_IDADE NU_IDADE 4.01448Em nenhuma das duas, TP_SEXO, nossa variável que indica o gênero, aparece. Todavia, agora a raça (TP_COR_RACA) surge, ainda que em posições baixas. Podemos plotar o efeito isolado de cada um desses preditores na nota de matemática. Vamos tentar plotar a renda no Rio e em São Gonçalo para ver como essa variável influencia no resultado final:
plot(boost.enemrio, i ="Q006", main = "Rio de Janeiro - Renda")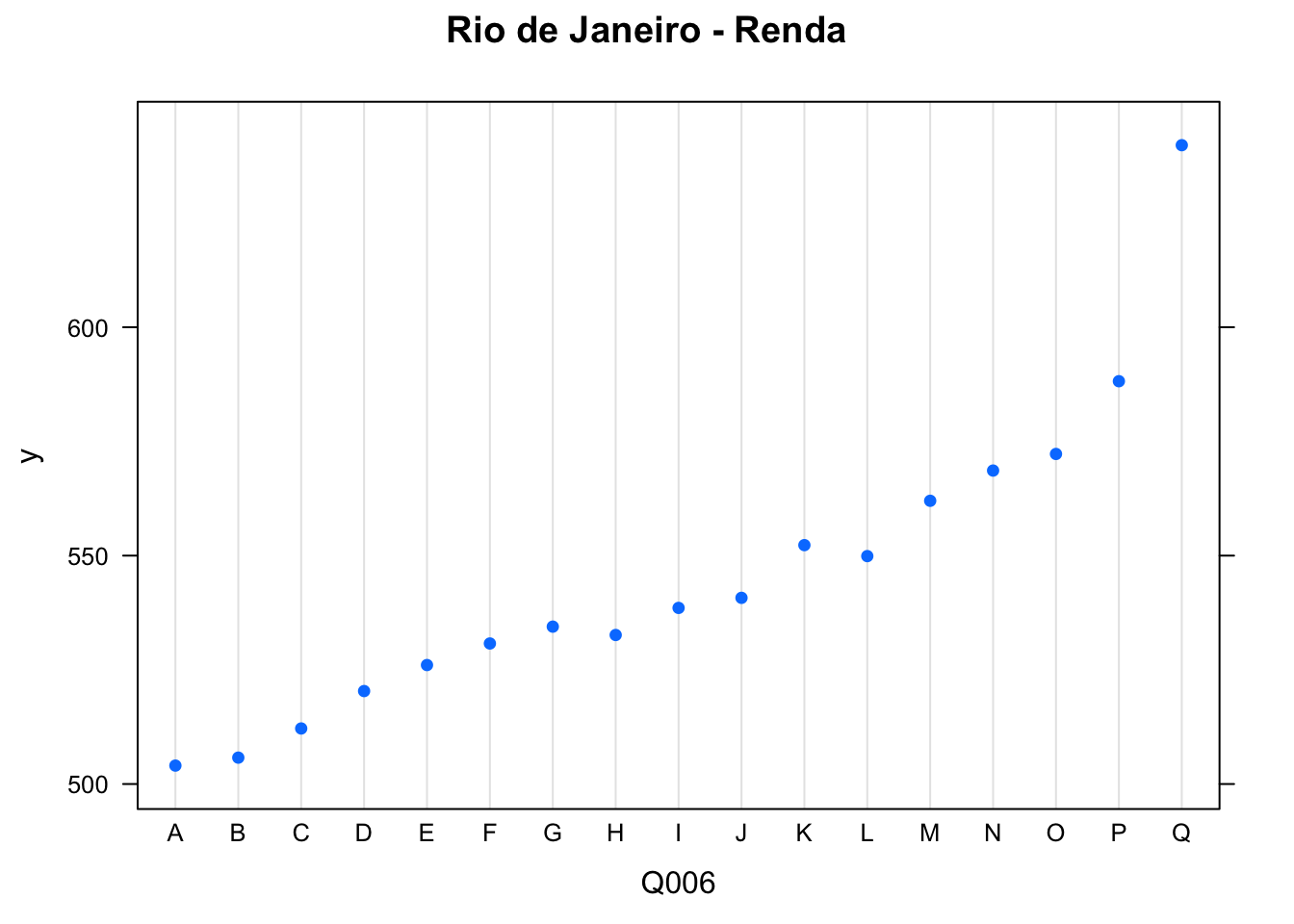
plot(boost.enemsg, i = "Q006", main = "São Gonçalo")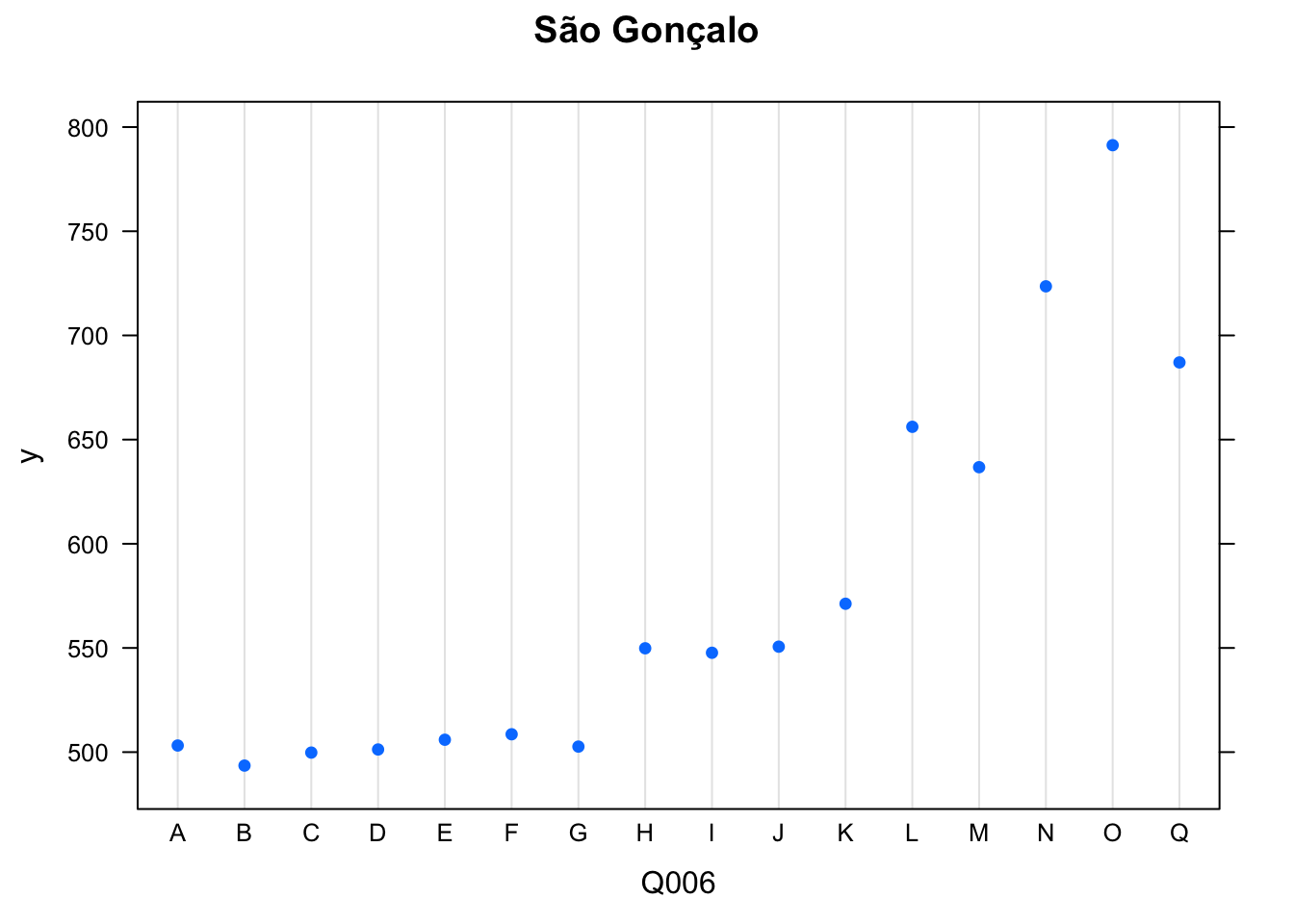
plot(boost.enemrio, i ="TP_COR_RACA", main = "Rio de Janeiro - Cor/Raça")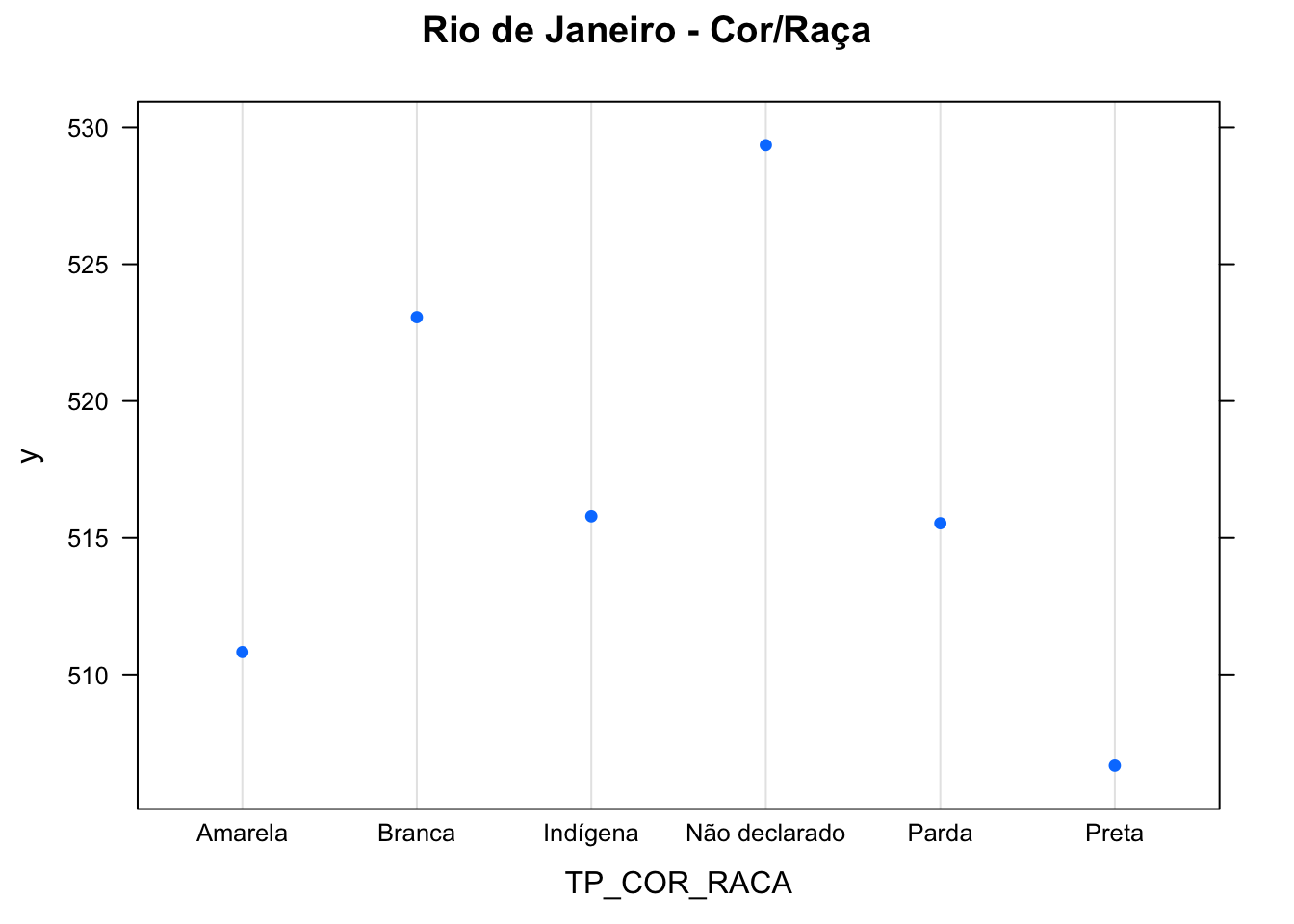
plot(boost.enemsg, i ="TP_COR_RACA", main = "São Gonçalo - Cor/Raça")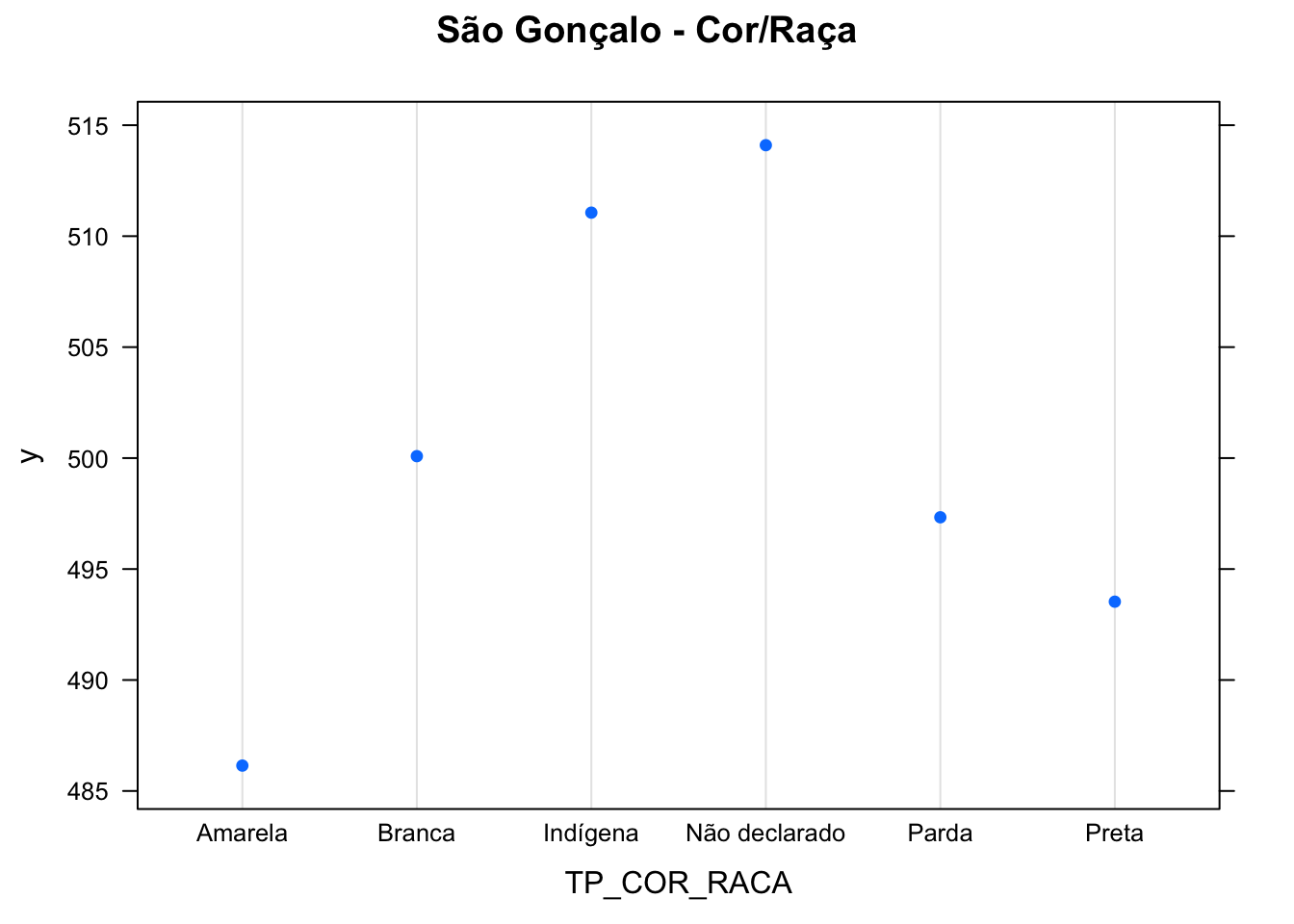
Percebe-se que em ambos quanto maior a renda, maior a nota, no Rio. Já em São Gonçalo, o mesmo se verifica com alguns degraus e, na última faixa de renda, “Q”, para rendas maiores de 19 mil reais, a nota cai, em comparação com “O”, que representa a renda famíliar entre R$ 11448,00 e R$ 14.310,00.
No que tange à cor/raça, percebemos que a população parda e preta tem um desempenho mais baixo em comparação à população branca, dada as opressões estruturais que atingem esses grupos, afetando o ensino e aumentando a desigualdade social.
Algumas conclusões
Algumas conclusões podem ser feitas a partir dos dados preliminares utilizados:
- a renda é sim um fator importantíssimo, junto com a escolaridade do pai e da mãe, na definição da nota de matemática do ENEM. Pais com maior formação tendem a criar filhos melhor instruídos, seja pela criação, seja pela escola;
- o tipo de escola e o fato de pertencer à rede estadual, municipal, federal ou privada não é tão relevante como se pensava, no âmbito do município do Rio e de São Gonçalo;
- a idade, dada por
NU_IDADE, parece ser relevante, o que pode ser explicado pelo seguinte pensamento: pessoas mais velhas já passaram pela formação escolar há mais tempo ou apresentam um déficit na mesma, mostrando maior dificuldade devido a isso; - o sexo não é relevante nas análises, mas uma possível justificativa (e que pode e deve ser investigada posteriormente) é que há uma diminuição na nota de mulheres pois em um considerável número de famílias, o trabalho doméstico não é igualmente dividido entre homense mulheres, restando às mulheres que dividam suas funções, como trabalho e estudo, com tais afazares. Isso diminui consideravelmente o rendimento escolar.
Quem quiser olhar o dicionário do banco, pode conferir aqui.
Bom pessoal, por hoje é só. Vimos como uma abordagem de árvores de decisão, somada a métodos como bagging, randomforests e boosting podem nos auxiliar no desenho de políticas públicas direcionadas para educação. Que critérios devem ser priorizados? Como poderíamos avaliar o impacto de uma política de renda, por exemplo, em um município? Que grupos devem ser alvo de políticas distributivas primeiro? Todas essas questões podem ser respondidas ou pelo menos iniciadas com os métodos vistos no post de hoje. Além disso, percebemos como a desigualdade afeta alunos de municípios próximos, e nos deixa refletindo como a situação em todo o país é distinta, exigindo cuidados específicos. Porém, um ponto é claro: é preciso democratizar cada vez mais o ensino público e o acesso à universidade, e não é tratando todos os casos como o mesmo (como no caso do atual governo, que propõe a adoção progressiva do ENEM Digital) que a desigualdade será solucionada.
Qualquer dúvida, correção ou sugestão pode ser encaminhada para matheus.pestana@iesp.uerj.br .